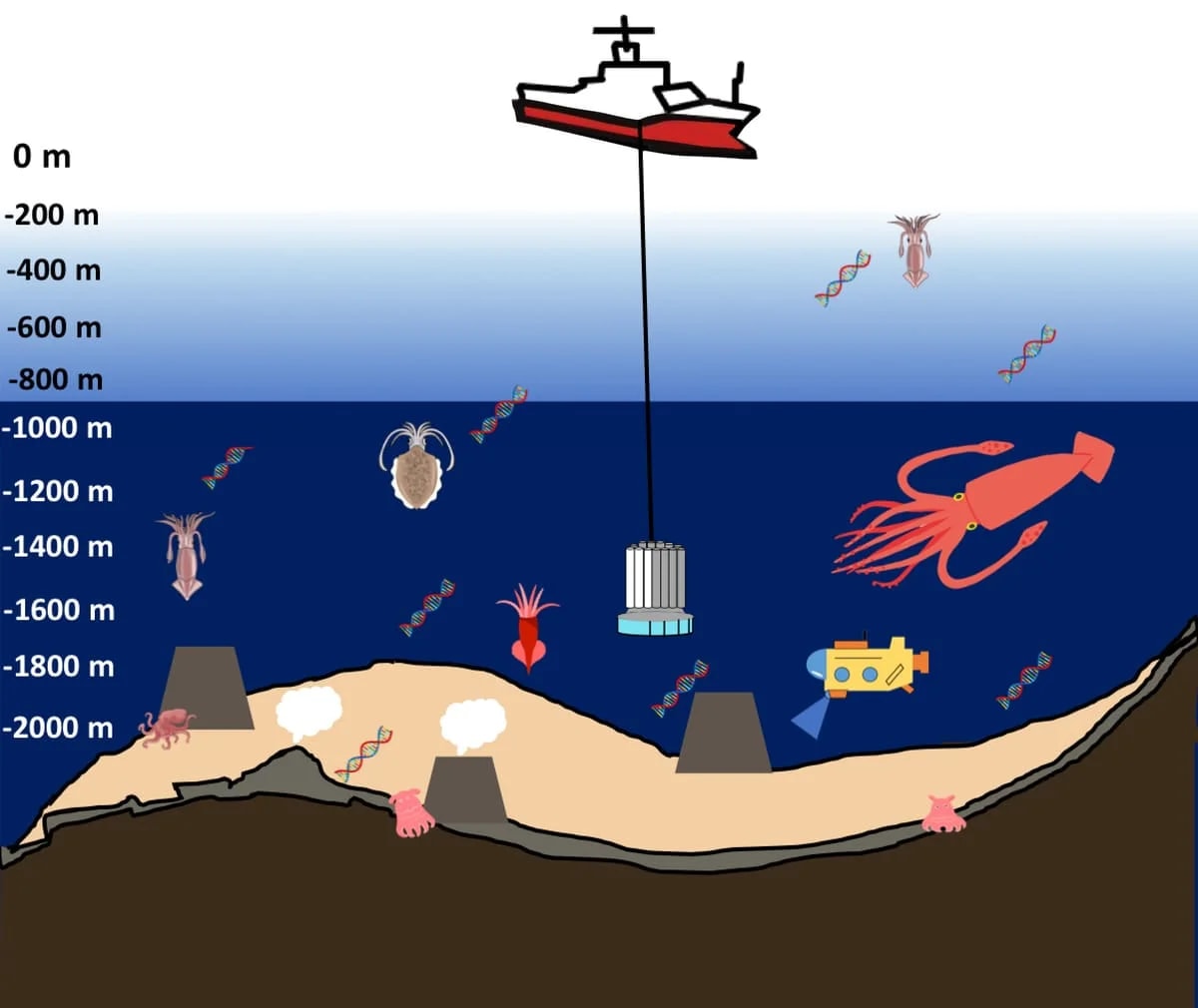
Un equipo de investigadores liderado por la Universidad de Kobedesarrolló un método eficaz para rastrear la presencia de calamares y pulpos en aguas profundassin necesidad de capturarlos directamente. La técnica, basada en la codificación de barras metabólica del ADN ambiental, permite identificar especies mediante fragmentos de material genético presentes en el agua, incluso a profundidades de hasta 2000 metros.
El estudio, publicado en la revista Marine Environmental Research, representa un avance significativo en el estudio de los cefalópodos, un grupo clave para los ecosistemas oceánicos que, debido a su movilidad y hábitats remotos, ha sido históricamente difícil de observar. Según el ecólogo marino WU Qianqian, autor principal del estudio, los cefalópodos juegan un rol esencial en la distribución de energía y nutrientes en la cadena trófica marina.
La técnica consiste en diseñar “cebadores” o sondas genéticas que actúan como cebos moleculares, capaces de capturar secuencias específicas de ADN pertenecientes a diferentes especies. El desafío, según Wu, fue lograr un equilibrio entre especificidad y generalidad: los cebadores deben identificar con precisión al grupo objetivo —en este caso los cefalópodos— sin excluir especies poco conocidas. Para ello, el equipo colaboró con la Agencia Japonesa de Ciencia y Tecnología Marina-Terrestre (JAMSTEC), que proporcionó acceso a muestras recolectadas desde la superficie hasta las profundidades oceánicas.
Los resultados fueron contundentes. Las sondas lograron detectar ADN de múltiples especies de calamares y pulpos, tanto en muestras simuladas como en agua real de mar profundo. En particular, la detección de ADN de pulpos exclusivamente en capas profundas coincide con su estilo de vida bentónico, solitario y oculto, lo que confirma la precisión del método. Además, el uso de fragmentos de ADN más largos —posible gracias a las bajas temperaturas del mar profundo— permitió una identificación más fiable de las especies presentes.
Este enfoque no solo evita la captura invasiva de organismos, sino que ofrece una herramienta valiosa para comprender patrones de distribución, ciclos de vida y comportamiento de especies difíciles de estudiar. Wu advierte, sin embargo, que aún quedan desafíos pendientes, como mejorar las bases de datos de ADN para reducir errores de identificación y ajustar los protocolos de muestreo según las características ecológicas de cada grupo.
El proyecto fue financiado por el Ministerio de Medio Ambiente de Japóny contó con la participación de investigadores de la Universidad de Kioto, el Museo de Historia Natural de Osaka, el Museo e Instituto de Historia Natural, la Fundación Churashima de Okinaway la propia JAMSTEC. Según los autores, esta tecnología podría servir de base para programas de monitoreo ecológico y conservación marina a gran escala.
“El mar profundo sigue siendo uno de los ecosistemas menos explorados del planeta. Nuestra técnica abre nuevas puertas para estudiar su biodiversidad de forma no invasiva y sostenible”, concluyó Wu. Con herramientas como esta, la ciencia se acerca un poco más a comprender la vida oculta en las profundidades del océano.
Referencia: Wu, Q. et al. (2025). Development of eDNA metabarcoding primers for cephalopods and their application to deep-sea samples. Marine Environmental Research. Disponible en: https://www.sciencedirect.com/science/article/abs/pii/S0141113625001515

 Iceebook
Iceebook